- Fixer plus rapidement les caractères
- La culture d’embryons immatures
- Faciliter l’introduction de nouveaux caractères
- Le sauvetage d’embryons interspécifiques
- L'hybridation somatique
- La mutagénèse
- La transgénèse : domaines d'application
- La transgénèse : stratégies
- La transgénèse : les étapes
- L'obtention d’une variété OGM
- Les méthodes récentes
- CRISPR-CAS9
- La biologie de synthèse
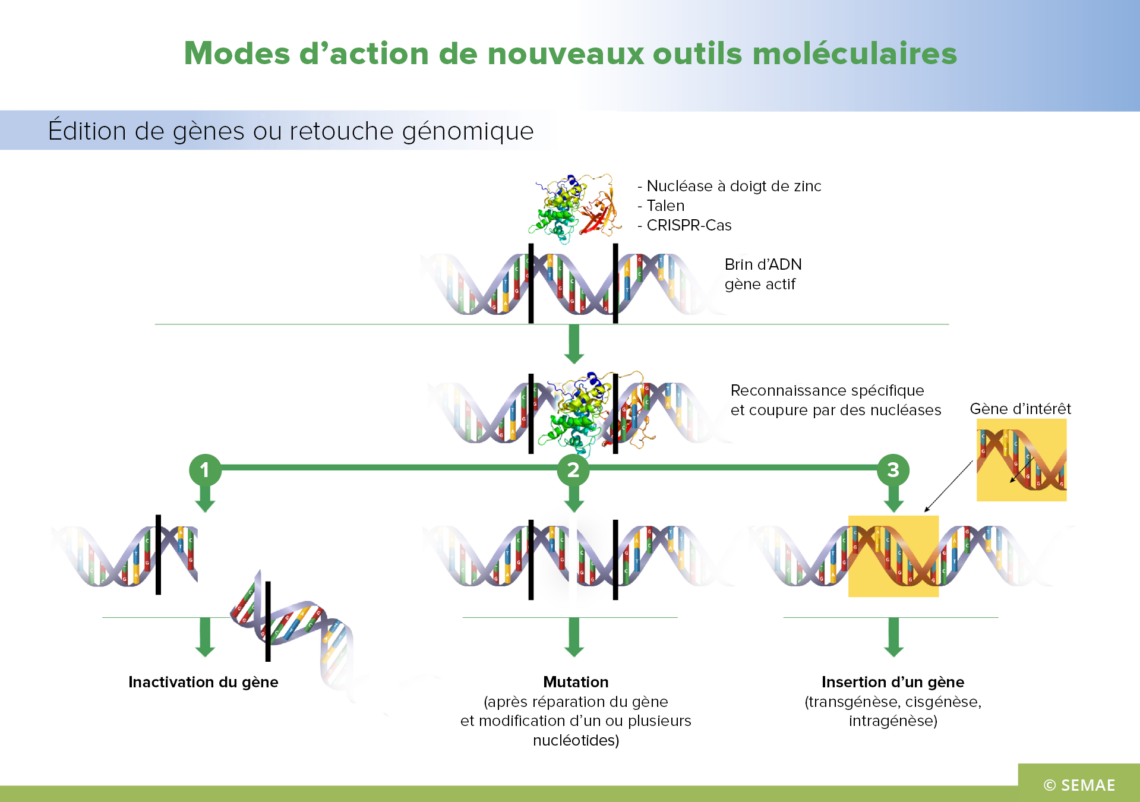
Edition du génome et retouche génétique par le système CRISPER-CAS9
MODES D’ACTION DE NOUVEAUX OUTILS MOLÉCULAIRES : ÉDITION DE GÈNES OU RETOUCHE GÉNÉTIQUE
La connaissance des génomes et surtout de leur fonctionnement a permis depuis une dizaine d’années de développer de nouveaux outils de sélection qui incluent d’une part, des modifications du génome (insertion, modification ou inhibition de gènes) et d’autre part, l’utilisation de protéines qui interfèrent avec l’expression du génome. C’est grâce aux progrès sur le génie génétique et la bio-informatique que ces outils ont vu le jour.
Utilisation de fragments d’ADN ou de protéines pour induire la mutagénèse ou la transgénèse
La fabrication d’oligonucléotides et de nucléases de synthèse est désormais courante dans les laboratoires de recherche. Ces fragments d’ADN (oligonucléotides) ou ces protéines (nucléases) permettent de produire des mutations du génome, ponctuelles, spécifiques et contrôlées.
Un oligonucléotide est un assemblage synthétique de nucléotides. Dans le cas de la mutagénèse ou de la transgénèse il est identique à celui d’une partie d’un gène que l’on veut modifier à l’exception d’un nucléotide « différent ». Introduit dans la cellule, il s’apparie avec l’ADN de la plante et sert de modèle pour le mécanisme naturel de réparation de l’ADN de la cellule. Celui-ci détecte le nucléotide « différent » et copie le changement voulu dans l’ADN de la plante. Ainsi, l’oligonucléotide n’est pas introduit dans le génome de la plante et est rapidement dégradé.
Les nucléases sont des enzymes (protéines) qui coupent les liaisons phosphodiesters entre deux nucléotides. Ces nucléases sont associées à des séquences qui reconnaissent spécifiquement des séquences d’ADN, et ainsi permettent de cibler des zones du génome où des modifications sont recherchées. La nucléase va couper l’ADN ciblé et le mécanisme de réparation naturel de la plante reconnait la coupure de l’ADN provoquée par la nucléase et la répare. L’objectif de cette technique est d’introduire un changement spécifique au niveau de la coupure : une délétion, une substitution ou l’addition d’un nombre plus ou moins important de nucléotides.
Les nucléases à doigt de zinc se caractérisent par la présence d’un ion de Zinc qui donne une forme tridimensionnelle particulière en ciseaux. Elles comprennent un domaine de liaison à l’ADN spécifique (qui induit la reconnaissance de l’ADN) et un domaine catalytique non spécifique (qui induit la coupure du double brin d’ADN). Elles agissent en se liant spécifiquement à une séquence précise d’ADN. Ces nucléases à doigt de zinc entraînent l’insertion ou la délétion ciblée d’un nucléotide, avec éventuellement pour conséquence l’apparition ou la disparition d’un caractère.
D’autres types de nucléases découvertes plus récemment peuvent être utilisés (méganucléases, TALENs). Elles utilisent toutes le même principe de reconnaissance spécifique d’une séquence d’ADN suivi d’une coupure de la liaison entre nucléotides. La modification engendrée peut être une délétion, une mutation ou l’insertion d’une séquence codante (transgène).
Les méganucléases se caractérisent par un site de reconnaissance de grande taille ce qui fait qu’il est généralement présent en un seul exemplaire dans un génome donné. Les méganucléases sont considérées comme les enzymes de restriction les plus spécifiques.
Les TALENs (Transcription Activator-Like Effector Nucleases) comportent aussi un domaine très spécifique se liant à l’ADN et un domaine catalytique. La séquence codante de la protéine TALE est insérée dans un plasmide d’expression (non intégré dans le génome) qui est introduit dans la cellule . La cellule produit ainsi la protéine TALE qui remplira sa fonction de clivage de l’ADN.
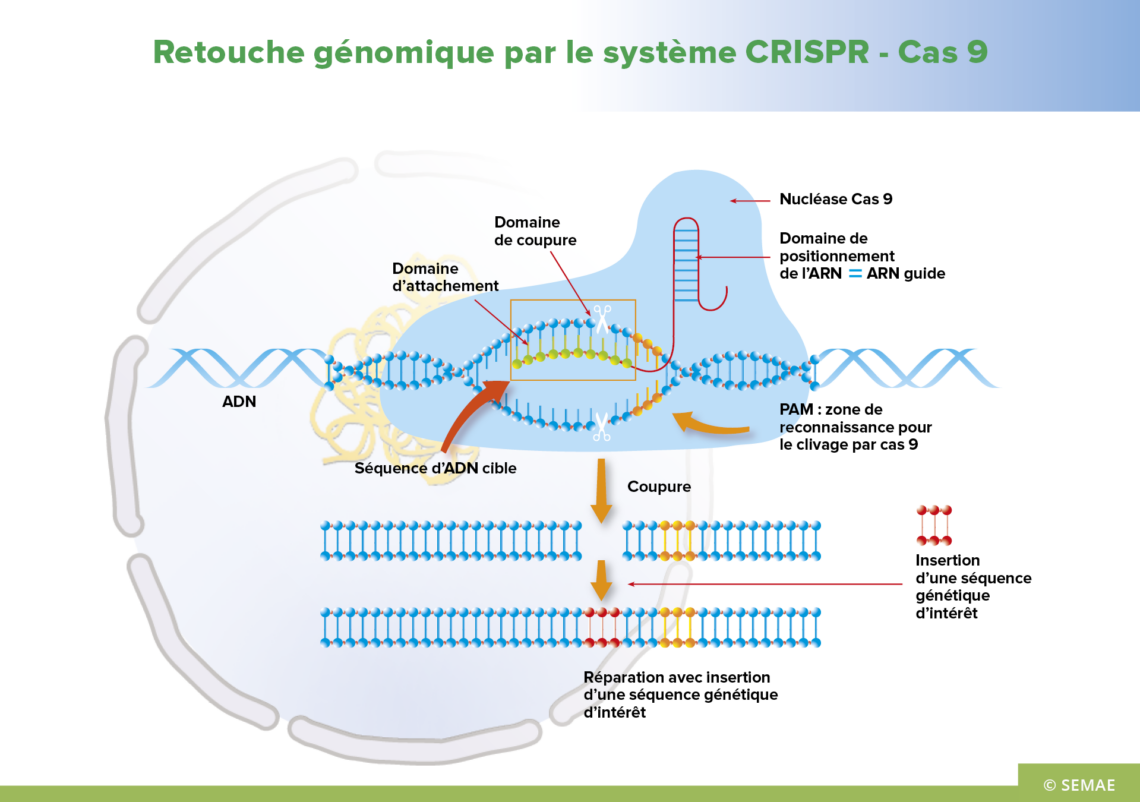
RETOUCHE GÉNÉTIQUE PAR LE SYSTÈME CRISPR-CAS9
Les nucléases à doigt de zinc se caractérisent par la présence d’un ion de Zinc qui donne une forme tridimensionnelle particulière en ciseaux. Elles comprennent un domaine de liaison à l’ADN spécifique (qui est responsable de la reconnaissance de l’ADN) et un domaine catalytique non spécifique (qui induit la coupure du double brin d’ADN). Le domaine de liaison à l’ADN est constitué par une succession de peptides reconnaissant chacun une séquence de trois paires de bases.
La spécificité d’une telle chaîne artificielle de peptides n’étant pas parfaite et la construction aléatoire, il faut en général plusieurs dizaines de ce type de nucléases pour obtenir une spécificité acceptable et il faut combiner deux nucléases à doigt de zinc car chacune ne coupe qu’un seul brin d’ADN.
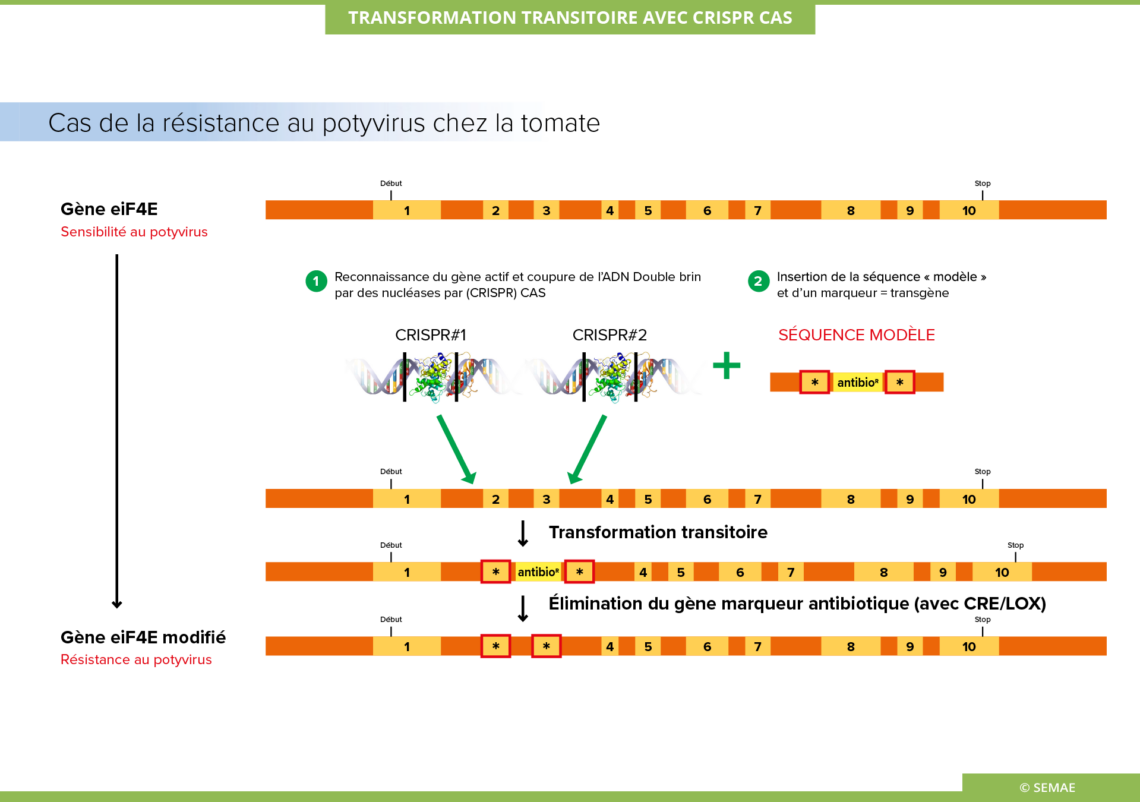
TRANSFORMATION TRANSITOIRE AVEC CRISPR-CAS
CRISPR-Cas9 est un outil d’ingénierie du génome couramment utilisé dans les laboratoires de recherche pour produire des ruptures ciblées du double brin d’ADN. Cas9 est une enzyme spécialisée, une endonucléase, qui associée à un ARN-guide est capable de couper l’ADN à un site déterminé par la séquence de cet ARN-guide.
Ces cassures induites, comme toute cassure de l’ADN, vont être réparées par la machinerie interne de réparation de la cellule, ce qui peut conduire à l’inactivation ou à la modification fine d’un gène, ou encore à l’insertion d’un transgène.
Dans le cas 2 du schéma, la modification de quelques nucléotides peut s’effectuer par recombinaison homologue à partir d’une séquence « modèle », pour induire un changement de fonction du gène ciblé. Cette technique de modification précise du génome a été utilisée par des équipes de l’INRAE pour prémunir la tomate contre les potyvirus.
Recherche de gènes récessifs qui confèrent une résistance au potyvirus chez la tomate
Parmi les mutants récessifs identifiés chez les plantes qui présentent une résistance aux virus, il y a ceux du gène « eiF4E1 » qui s’avère essentiel pour la diffusion du virus, tant pour pour la phase d’amplification de son génome que celle de son encapsidation qui protège son ARN. De tels mutants chez la tomate et le melon ont permis d’obtenir une certaine résistance mais avec un spectre étroit et surtout au prix d’une réduction de croissance et de fertilité de la plante. D’autres mutations naturelles de ce gène ont été observées chez le poivron qui confèrent une résistance plus large et sans effets négatifs par ailleurs : dans ce cas, ce sont deux acides aminés de la protéine eIF4E qui sont modifiés.
L’objectif du projet GENIUS de l’INRAE a été de modifier le gène de la tomate selon le modèle du gène de résistance du poivron. Ce projet a donc consisté à tester deux outils TALEN et CRISPR-Cas9 pour induire chez la tomate cette résistance aux potyvirus à plus large spectre, beaucoup plus difficile à contourner.
L’utilisation de CRISPR-Cas dans le cadre du projet GENIUS
Les chercheurs ont d’abord testé l’efficacité de coupures du gène eiF4E1 à l’endroit désiré à l’aide de différents « ciseaux moléculaires » TALENs et CRISPRs. Le système CRISPR-Cas s’est avéré l’outil le plus efficace et le moins cher.
Ils ont construit en parallèle une séquence homologue au gène ciblé de tomate (elF4E1) contenant les deux mutations désirées permettant de modifier les deux AA impliqués dans la résistance au potyvirus chez le poivron et un gène marqueur (antibiotique) pour suivre la transformation effectuée par biolistique. Celle-ci consiste en l’utilisation de microbilles de métal enrobées d’ADN (billes d’or ou de tungstène d’un micromètre de diamètre) qui sont projetées à très grande vitesse sur les cellules à modifier afin de traverser leur paroi. La transformation est dite transitoire car les outils CRISPR (endonucléase et ARN guide) sont présents de manière transitoire avant d’être détruits par les systèmes de « nettoyage » de la cellule.
Les chercheurs ont sélectionné les plantes contenant l’insertion complète et les ont testées sous serre.
Ils cherchent désormais le meilleur système de recombinaison pour éliminer le gène marqueur antibiotique qui a permis de faire le tri des transformations obtenues.
A terme, la transformation va permettre d’obtenir des plantules semblables à des plantules mutées naturellement ou par des agents mutagènes. Elles ne contiennent aucun transgène et ne sont différentes que sur 2 nucléotides du gène eIF4E1 permettant d’obtenir la même résistance au potyvirus que celle qui existe chez le poivron.
A chaque étape, les chercheurs transfèrent les plantules en serre et les graines des plantes autofécondées sont récoltées. Celles qui sont homozygotes pour la mutation souhaitée sont conservées et multipliées de nouveau sous serre.
Les tests avec le potyvirus en zone confinée permettent de vérifier si la mutation obtenue a le même effet que l’extinction du gène eiF4E1 pour les différentes plantes régénérées et si celles-ci présentent une croissance normale. Des essais au champ seront impératifs pour valider le projet scientifique avant toute phase de mise sur le marché.
Résistance au potyvirus
Les potyvirus sont des virus très répandus et très nombreux chez les végétaux. Ils représentent le tiers des virus recensés. Transmis essentiellement par les pucerons, ils provoquent de graves pertes dans les cultures de fruits et légumes. Le génome du potyvirus est constitué d’une molécule d’ARN simple brin. Après pénétration dans la cellule et amplification de son génome par la plante, le virus va à la fois interférer avec le système immunitaire pour le déprimer et infecter progressivement les cellules de la plante cible.
Certaines plantes sont très infectées et les dégâts se caractérisent alors par un rabougrissement de la plante et une perte conséquente de récoltes. D’autres plantes le sont moins, voire pas du tout. Certaines plantes présentent en fait des phénotypes de récupération (après une phase initiale de sensibilité, les plantes récupèrent et deviennent résistantes) alors que d’autres variétés ne permettent même pas au virus de se multiplier (résistance extrême la plus intéressante en agriculture).
Les axes de recherche pour lutter contre les potyvirus
Au niveau du virus :
- Caractérisation des facteurs viraux de la pathogénicité des potyvirus pour identifier rapidement les souches les plus pathogènes et lutter plus rapidement contre elles.
- Compréhension des mécanismes du virus pour contrer les systèmes de défense de la plante. Les plantes mutantes ou transformées « insensibles » au virus fourniront des résistances à large spectre.
Au niveau de la plante :
- Identification des facteurs -hôte nécessaires à la multiplication virale et identification de plantes mutantes pour ces facteurs. Les interactions plantes -virus indispensables au développement du virus seront neutralisées. Plusieurs gènes, voire des régions de chromosomes (QTL), peuvent conférer des résistances à une plante.
- Transformation des plantes pour induire une résistance durablement efficace, par transgénèse (cas d’une pastèque GM autorisée au Canada et aux USA) ou par CRISPR Cas 9 (cas du concombre GM autorisé aux USA, ou encore à l’état de la recherche pour la tomate)
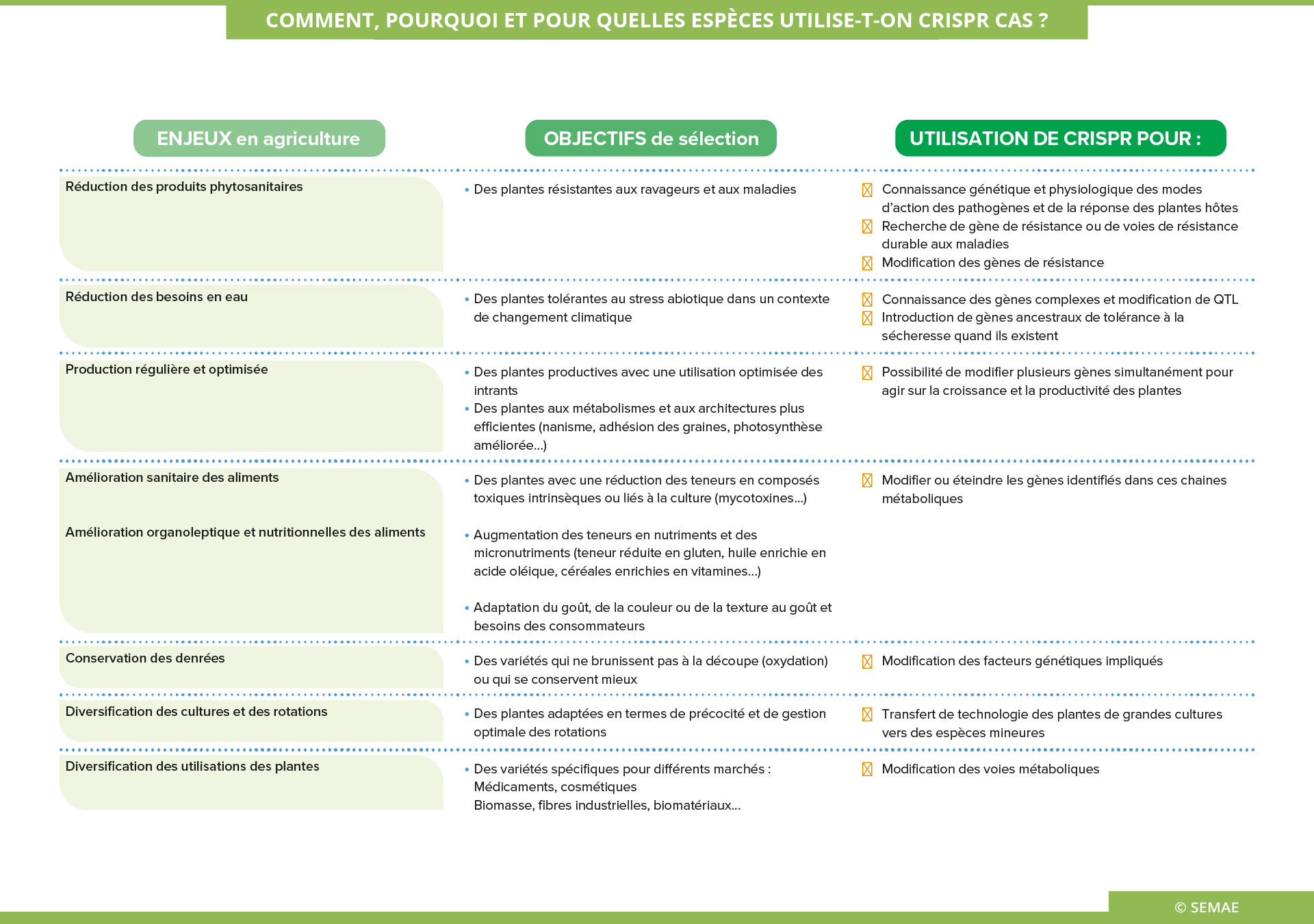
COMMENT, POURQUOI ET POUR QUELLES ESPÈCES UTILISE-T-ON CRISPR-CAS ?
La connaissance fine du génome et la compréhension des mécanismes physiologiques révolutionnent la façon de guérir les humains de maladies génétiques ou de dysfonctionnement cellulaire comme le cancer, ou d’améliorer les qualités nutritionnelles et sanitaires des plantes. La paire de ciseaux moléculaire CRISPR-Cas s’avère, d’un point de vue conceptuel, facile et précise, mais son utilisation pour des objectifs concrets en santé humaine ou en amélioration des plantes nécessite encore beaucoup de recherches.
Rappel du mécanisme CRISPR-Cas
CRISPR-Cas est un complexe formé d’une protéine Cas9, capable de couper l’ADN et d’une séquence d’ARN (sgRNA) qui permet la reconnaissance d’une séquence d’ADN ciblée. Donc pour pouvoir utiliser CRISPR-Cas, il faut des connaissances préalables sur le ou les gènes que l’on veut modifier et avoir un objectif d’amélioration précis. Il faut également maîtriser l’ingénierie cellulaire (dans l’espèce et le génotype d’intérêt) car la modification de l’ADN se fait lors de la réparation de la cassure par la cellule végétale. Une réparation parfaite (la très grande majorité des cas de cassure spontanée) n’apporte aucun changement.
Les modifications possibles :
- Une coupure – simple – sans réparation induit une mutation qui rend généralement le gène non fonctionnel.
- Une réparation avec un « modèle » (une séquence homologue modifiée) induit une modification du gène et de sa fonction. Le remplacement d’allèles n’est pas effectué en routine actuellement. Il est testé par exemple pour lutter contre un potyvirus de la tomate. Cette méthode peut élargir de manière ciblée la base génétique d’une plante.
- Une réparation avec un modèle contenant de l’ADN étranger induit une insertion ciblée d’un transgène (transgénèse).
L’utilisation de CRISPR-Cas reste difficile pour de nombreuses plantes et la probabilité de modification varie suivant les objectifs d’amélioration
La machinerie de réparation cellulaire est très efficace. Pour obtenir une modification sur le génome, il faut la provoquer. La recombinaison homologue pour induire la modification fonctionnelle d’allèles ou l’introduction d’un transgène se fait actuellement avec des taux d’efficacité de moins de 1%.
CRISPR-Cas a montré son efficacité pour induire des mutations de type « inactivation ciblée de gène » sur le blé (résistance à l’oïdium), le tabac (résistance à un virus), le concombre (résistance à trois virus), la vigne (résistance à des champignons), l’orge (nanisme), la tomate (ramification ou taille du fruit), le colza (meilleure adhésion des graines), le maïs et la pomme de terre (amidon sans amylose), le blé (faible teneur en gluten)… Un champignon et une pomme qui ne brunissent plus lors de la découpe sont déjà au stade de la commercialisation en Amérique du Nord.
Beaucoup d’expérimentations sont en cours pour modifier un ou plusieurs allèles sans introduction de gène étranger dans le produit final.
L’édition de gènes sensu stricto demeure techniquement délicate.
Les promesses et les limites de CRISPR-Cas
CRISPR-Cas peut être utilisé pour une large gamme de traits, sachant que les traits sous dominance d’un seul gène (de type résistance aux maladies) sont plus accessibles que les traits polygéniques (comme la tolérance à la sécheresse).
CRISPR-Cas est plus précis et plus rapide que la sélection de mutations spontanées ou induites, car il reconnaît une séquence prédéterminée. On peut agir simultanément sur deux ou plusieurs gènes, sur un ou plusieurs sites d’un gène, impliqués dans un trait à améliorer. Il induit peu ou pas de modifications ailleurs dans le génome chez les plantes.
CRISPR-Cas peut fournir des allèles absents de la variabilité naturelle de l’espèce. Il peut permettre une domestication accélérée en introduisant les gènes majeurs qui ont influencé la domestication d’une espèce. (par exemple les 5 gènes majeurs pour la téosinte qui est l’ancêtre du maïs). A l’inverse, il peut permettre de réintroduire un état ancestral (pour rechercher de la vigueur perdue ou des gènes de tolérance à des stress). CRISPR-Cas peut donc créer de la variabilité originale pour les sélectionneurs.
CRISPR-Cas est une combinaison unique pour chaque transformation souhaitée. L’aspect réglementaire de son utilisation n’est pas encore clairement défini en Europe (en date de juin 2018).
Il existe une effervescence d’innovations autour de CRISPR-Cas, que ce soit pour créer des collections de mutants, pour obtenir des mutations ciblées multiples, ou encore pour réguler des gènes. Ainsi un complexe nommé dCas9 permet de reconnaître un gène et d’en réguler son activité. Il n’existe donc plus un seul complexe mais de nombreux complexes qui peuvent agir sur le génome des plantes, des animaux et des hommes. Leur utilisation et le type de transformation attendue devront donc être analysées au cas par cas.