Les marqueurs moléculaires
LES MARQUEURS RFLP
Caractéristiques du polymorphisme
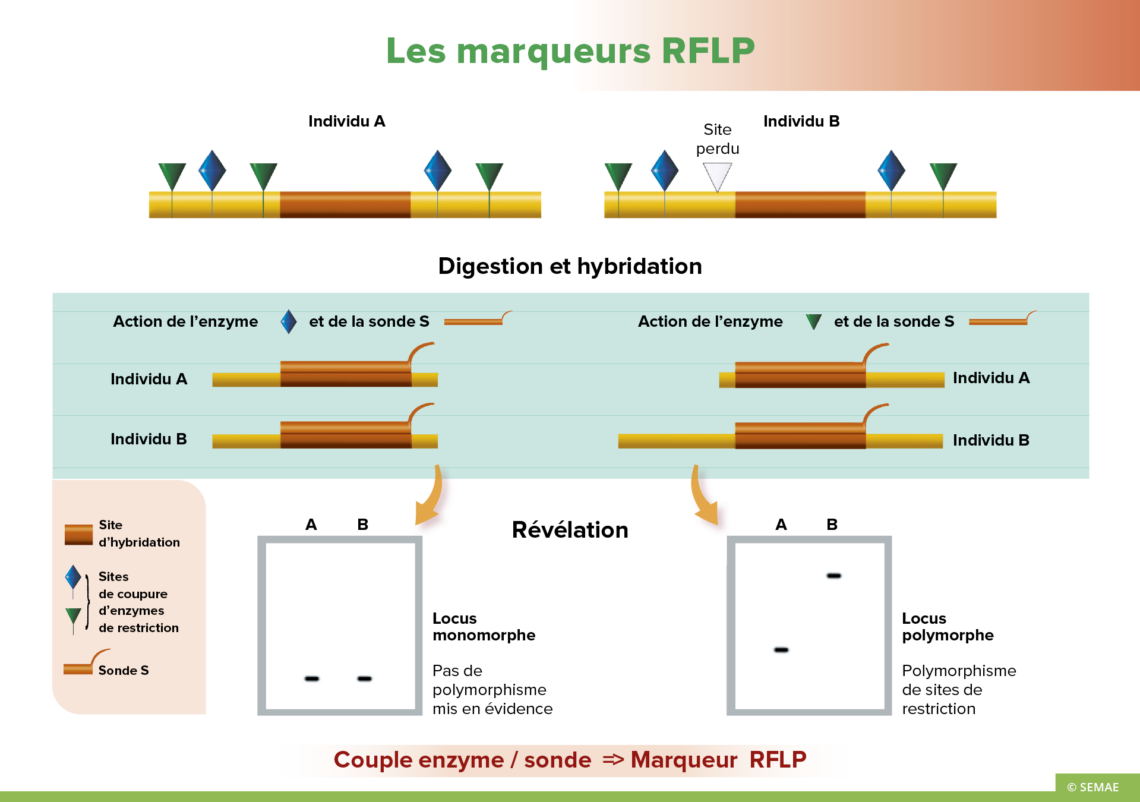
Comparons deux individus A et B. Leur ADN sera digéré séparément par des enzymes de restriction données, puis hybridé par une sonde S.
La digestion avec l’enzyme ♦ donne des fragments de restriction identiques pour les deux individus.
Les deux profils révélés après électrophorèse ne permettent pas de les distinguer.
Avec ce couple enzyme ♦ – sonde S, aucun polymorphisme n’est mis en évidence.
En revanche, pour l’enzyme ▼, l’individu B présente une mutation au niveau d’un site de restriction, entraînant la perte de ce site. Ainsi, par digestion, l’individu A donne un fragment plus petit que celui de l’individu B.
On révèle le polymorphisme entre les deux individus : un fragment rapide pour A et un plus lent pour B.
Ce couple enzyme ▼ – sonde S révèle un polymorphisme.
Création de marqueurs
C’est le couple enzyme/sonde qui constitue le marqueur. Le fragment d’ADN utilisé comme sonde révèle un locus polymorphe ou monomorphe.
Les enzymes de restriction permettent de découper l’ADN génomique et ainsi de visualiser le nombre d’allèles détectables, dans une population, à un locus donné. Chez le maïs, 95% des locus sont polymorphes, alors que chez le blé, plante autogame, 5 à 10% des locus seulement sont polymorphes.
Utilisation de la technique
Cette technique de marquage moléculaire a été très utilisée, car elle fournit des profils peu complexes permettant de caractériser l’empreinte génétique d’une plante ou de construire une carte génétique. Elle est fiable, les résultats observés peuvent être répétés ; elle est toutefois progressivement abandonnée au profit de techniques plus rapides à mettre en œuvre, et souvent basée sur la PCR.
LA TECHNIQUE RFLP
Polymorphisme de longueur des fragments de restriction (Restriction Fragment Length Polymorphism)
Toute modification des séquences d’ADN (mutation, addition, délétion) réorganise fréquemment les sites de restriction. Lors de l’action d’enzymes de restriction, la taille des fragments de restriction est alors modifiée : on observe un polymorphisme.
Les étapes de la technique
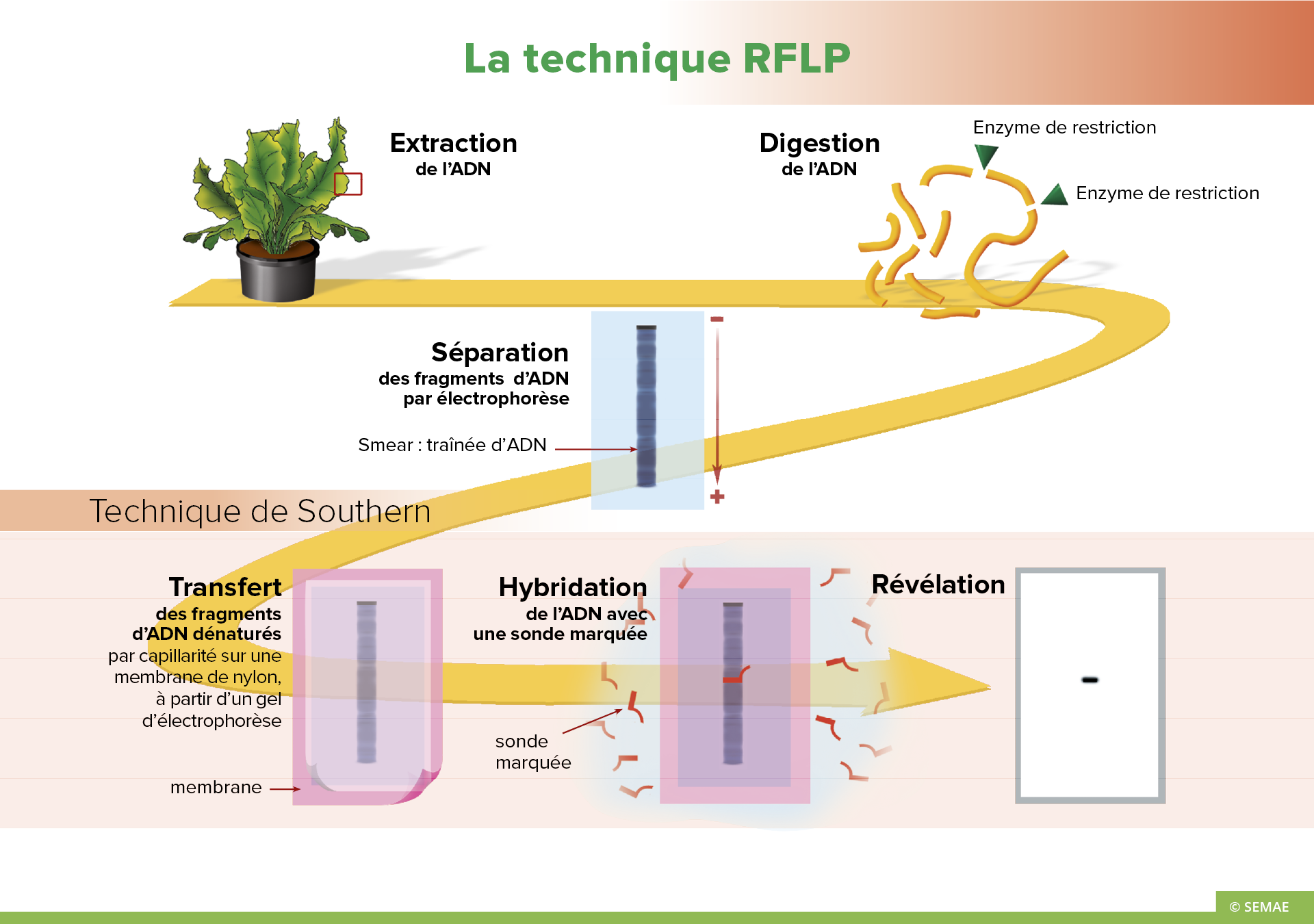
1. L’ADN de la plante est extrait.
2. Il est soumis à une digestion par une ou plusieurs enzymes de restriction. La taille des fragments obtenus est dépendante des enzymes utilisées.
3. Les fragments sont ensuite séparés selon leur taille par électrophorèse. Lors de la digestion de l’ADN génomique, on visualise sur le gel une traînée appelée « smear », car il y a un grand nombre de fragments de tailles différentes et impossibles à séparer.
4. L’ADN est transféré par capillarité sur une membrane de nylon où il est dénaturé. Cette technique de transfert permet de conserver la position relative des fragments d’ADN.
5. Cette membrane est mise en contact avec une solution contenant une sonde marquée soit par des isotopes, soit chimiquement. Cette sonde s’hybride alors avec le ou les fragments d’ADN avec lesquels elle présente une homologie complémentaire.
6. La position de l’hybridation est révélée en plaçant la membrane au contact d’un film sensible, ou en réalisant une réaction enzymatique colorée (selon le type de sonde utilisé).
L’extraction, la digestion, le transfert, l’hybridation et la révélation constituent la technique de Southern.
LES MARQUEURS MICROSATELLITES
Polymorphisme de nombre d’unités de répétition
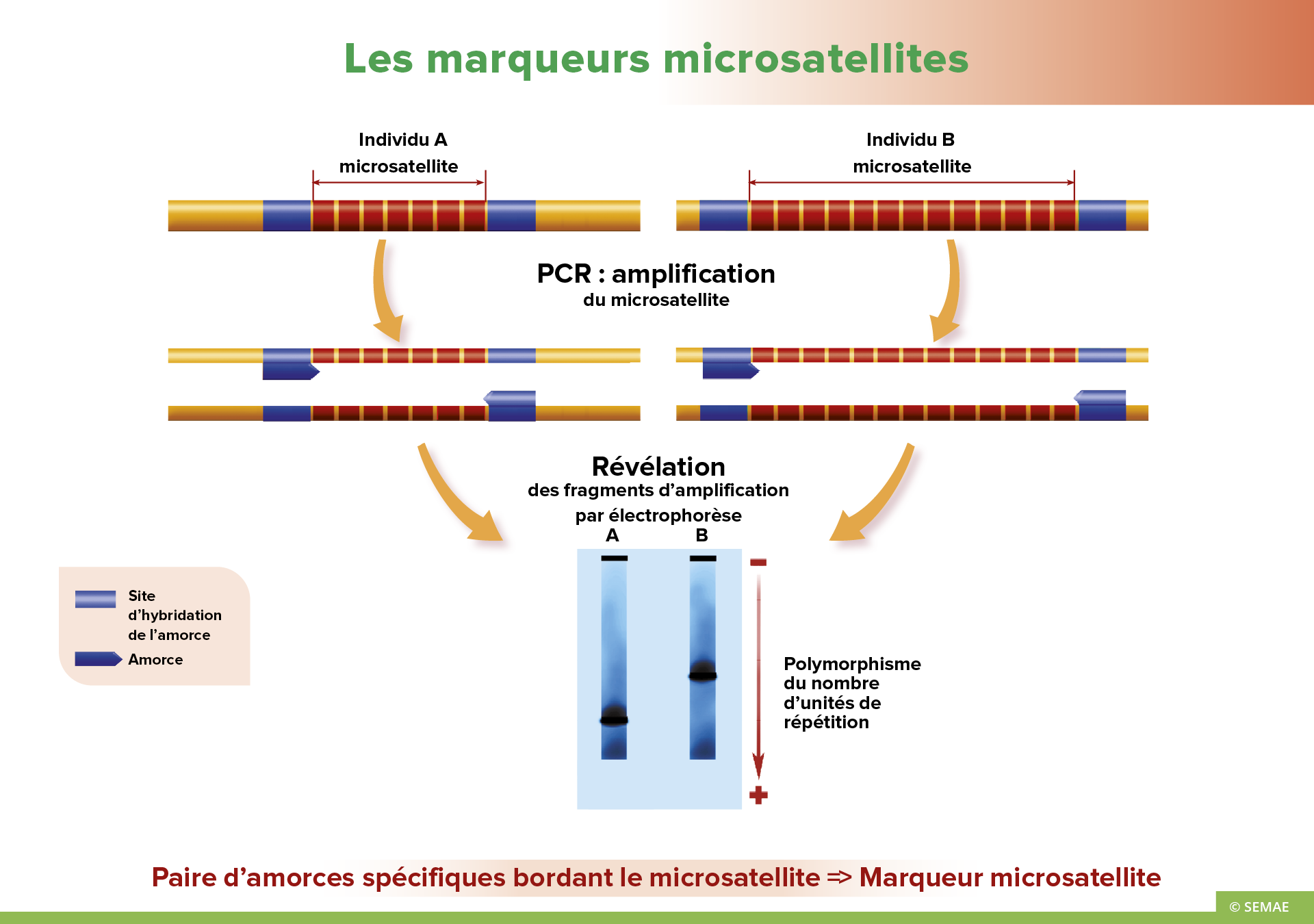
Sur le génome, il existe des séquences constituées d’unités répétées de 1 à 4 nucléotides. Ce sont les microsatellites. Les plus courants sont (A)n’ (TC)n’ (TAT)n et (GATA)n’, les valeurs de n pouvant aller de quelques unités à plusieurs dizaines. On parle de séquences répétées en tandem ou SSR (Simple Sequence Repeats). L’intérêt de ces microsatellites réside dans leur polymorphisme. Celui-ci repose sur la variation du nombre d’unités de répétition, constituant le microsatellite.
Les étapes de la technique
La technique de PCR est utilisée pour révéler le polymorphisme des microsatellites. Une paire d’amorces spécifiques des bordures droite et gauche d’un microsatellite est utilisée pour amplifier le même microsatellite chez différents individus. En effet, chaque microsatellite est bordé par des séquences uniques qui lui sont propres.
Les fragments d’amplification sont ensuite révélés par électrophorèse. Un individu B, possédant plus d’unités de répétition que A, a un produit d’amplification distinct et qui migre plus lentement que A.
Création de marqueurs
C’est la paire d’amorces spécifiques des bordures droite et gauche du microsatellite qui constitue le marqueur.
Utilisation de la technique
Il faut connaître, synthétiser et tester les amorces bordant le microsatellite. Cette technique est simple d’utilisation car repose simplement sur une PCR. Elle permet de développer de nombreux marqueurs, notamment pour le maïs ou le colza. Toutefois, elle n’est pas applicable à toutes les espèces, la tomate par exemple ne possède pas de polymorphisme pour les microsatellites.
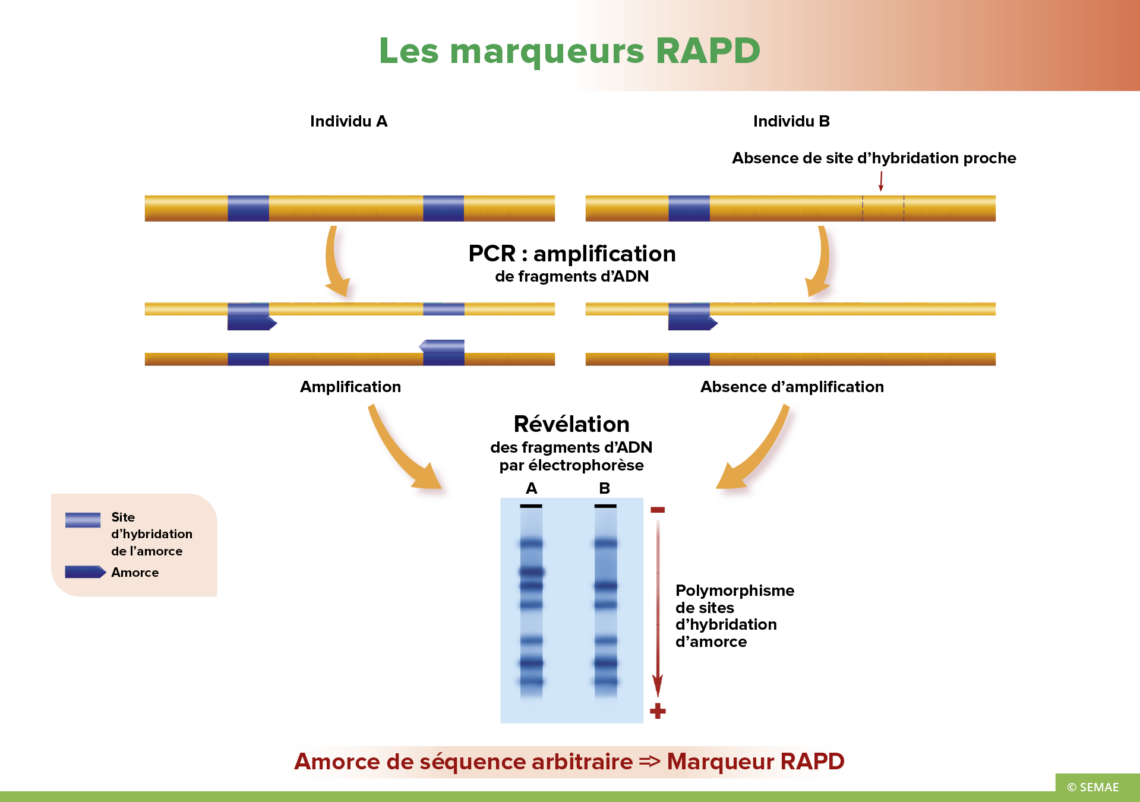
LES MARQUEURS RAPD
ADN polymorphe amplifié au hasard (Random Amplified Polymorphic DNA)
Cette technique consiste à réaliser une PCR en utilisant une amorce courte d’une dizaine de nucléotides, de séquence arbitraire. Cette amorce va s’hybrider au hasard dans le génome. Si deux sites d’hybridation sont proches et sur les deux brins d’ADN, il y aura amplification, c’est le cas de l’individu A. En revanche, si ces deux sites sont trop éloignés, il ne peut y avoir amplification, cas de l’individu B. Ainsi, on observe la présence d’une bande supplémentaire sur le gel pour A et l’absence pour B de cette même bande. Le polymorphisme révélé est un polymorphisme de sites d’hybridation d’amorce. Les amorces constituent donc les marqueurs. Pour l’ensemble du génome, une dizaine de fragments sont amplifiés en moyenne puis séparés par électrophorèse.
Utilisation de la technique
Cette méthode ne nécessite pas de digestion par une enzyme de restriction, pas de transfert sur une membrane, pas de préparation de sonde radioactive. Elle est donc rapide et d’une faible technicité. Toutefois, sa reproductibilité est difficile à obtenir. En effet, l’amplification obtenue dépend beaucoup des conditions de réalisation de la PCR. Ainsi les résultats sont difficilement reproductibles d’un laboratoire à un autre.
Elle est utilisée lors d’analyses préliminaires rapides, par exemple la mise en évidence de quelques marqueurs au voisinage d’un gène d’intérêt.
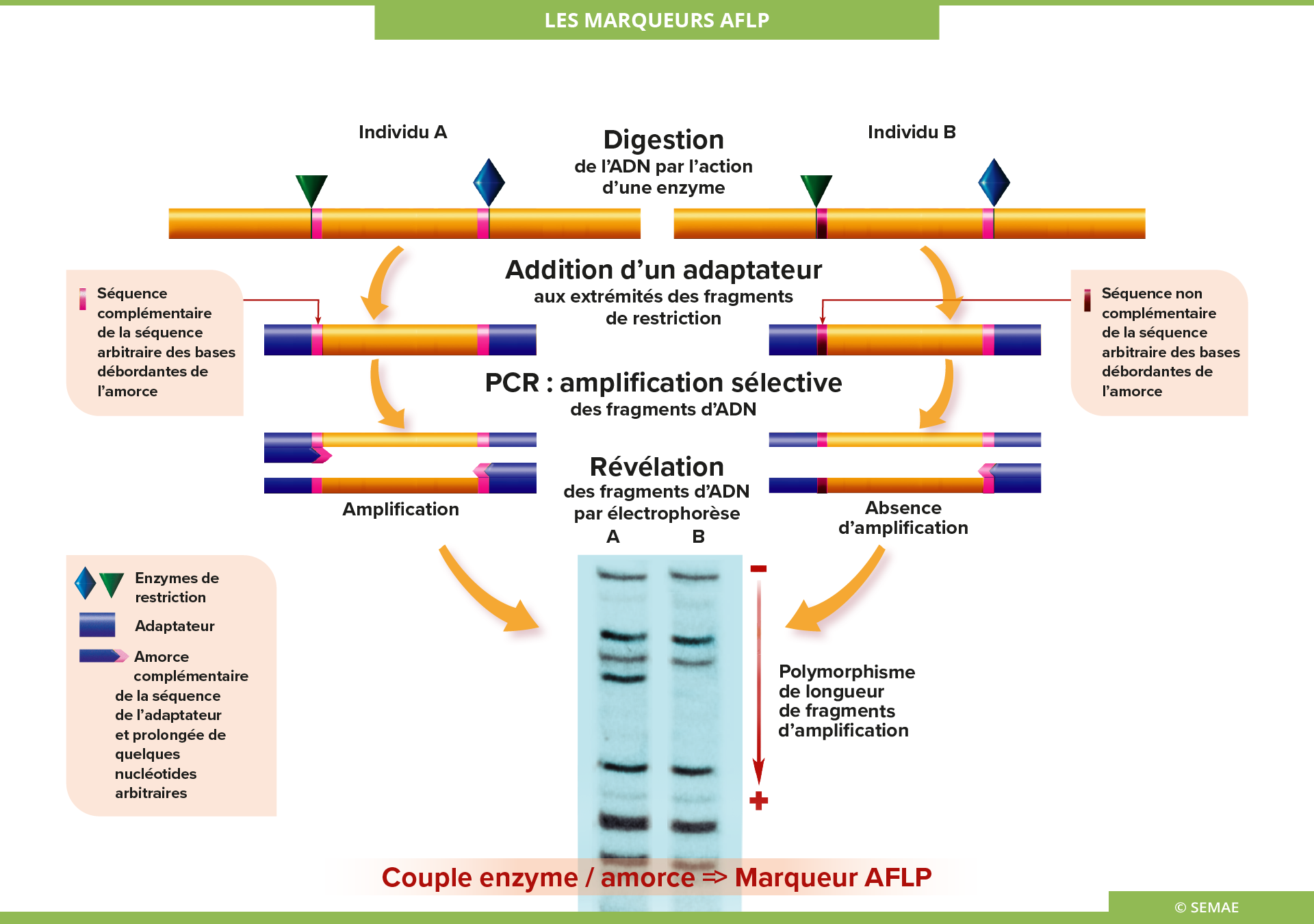
LES MARQUEURS AFLP
Polymorphisme de longueur des fragments d’amplification (Amplification Fragment Length Polymorphism)
Cette technique est fondée sur la mise en évidence conjointe de polymorphisme de site de restriction et de polymorphisme d’hybridation d’une amorce de séquence arbitraire.
Les étapes de la technique
L’ADN de la plante est soumis à une digestion par des enzymes de restriction. Les tailles des fragments obtenus sont dépendantes des enzymes utilisées.
Ensuite, il y a addition aux extrémités des fragments de restriction d’adaptateurs nucléotidiques spécifiques des enzymes de restriction utilisées. Ils sont de séquences connues.
Les fragments sont ensuite amplifiés par PCR. On utilise comme amorce un oligonucléotide complémentaire de la séquence de l’adaptateur, prolongé de quelques nucléotides arbitraires (de 1 à 3) appelés bases débordantes. Seuls sont amplifiés les fragments possédant les bases complémentaires de ces bases arbitraires. Il s’agit donc d’amorces sélectives permettant de réduire le nombre de fragments amplifiés à une centaine : sans ces séquences débordantes, il y aurait amplification de milliers de fragments.
Les bandes sont visualisées par électrophorèse.
Création de marqueurs
C’est la combinaison enzyme de restriction/amorce qui permet de révéler le polymorphisme entre les individus. Celle-ci constitue le marqueur AFLP. Le locus mis en évidence dépend de la séquence du site de l’enzyme de restriction et des bases arbitraires. Il existe de très nombreuses combinaisons enzyme/amorce. Il existe plusieurs centaines d’enzymes de restriction, mais une dizaine est généralement utilisée.
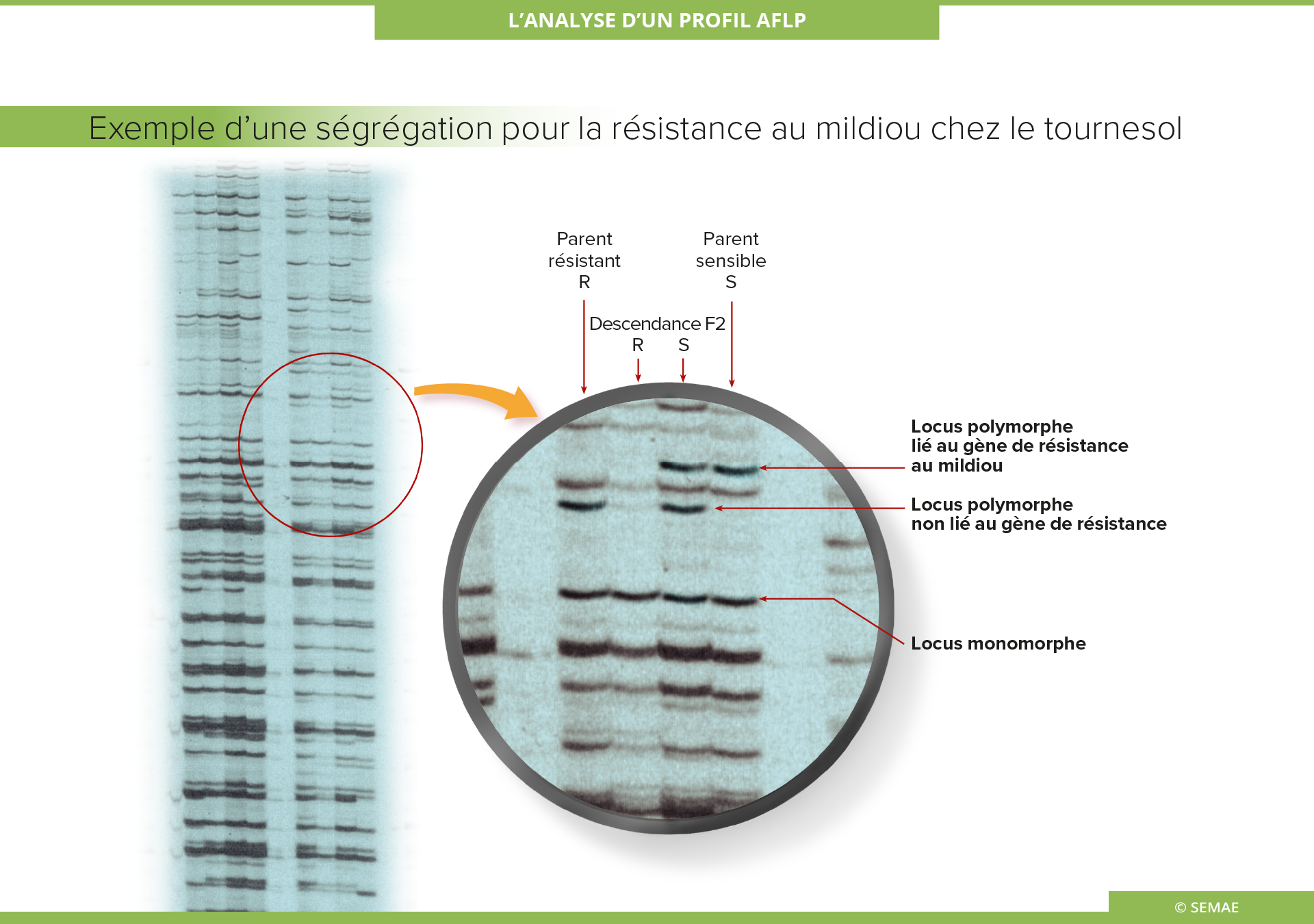
L’ANALYSE D’UN PROFIL AFLP
Caractéristique du polymorphisme
Sur les quatre pistes est mise en évidence la ségrégation pour la résistance du tournesol au mildiou. Sur la piste de gauche, c’est le parent résistant (R), sur la piste de droite le parent sensible (S). Sur les deux pistes centrales figurent les individus F2 : la deuxième piste en partant de la gauche correspond à l’ADN en mélange des descendants F2 résistants et la troisième piste à l’ADN en mélange des descendants F2 sensibles.
Parmi la centaine de fragments d’amplification séparés par électrophorèse, on peut visualiser des locus polymorphes. Il est notamment possible de mettre en évidence des bandes présentes chez le parent sensible et absentes chez le résistant, et inversement. Cette distinction se retrouve également dans la descendance. Ces locus sont donc liés au gène de résistance par absence ou présence de bande. Les autres locus soit ne révèlent pas de polymorphisme, soit révèlent des marqueurs polymorphes mais ne permettent pas de faire la distinction entre les individus sensibles et les individus résistants, ils ne sont donc pas liés à la résistance.
Utilisation de la technique
Elle est utilisée notamment pour la sélection de lignées, et pour la saturation d’une région du génome au voisinage d’un gène en vue de son clonage, c’est-à-dire pour positionner dans cette région un grand nombre de marqueurs.
LA COMBINAISON DES MARQUEURS
Les puces à ADN
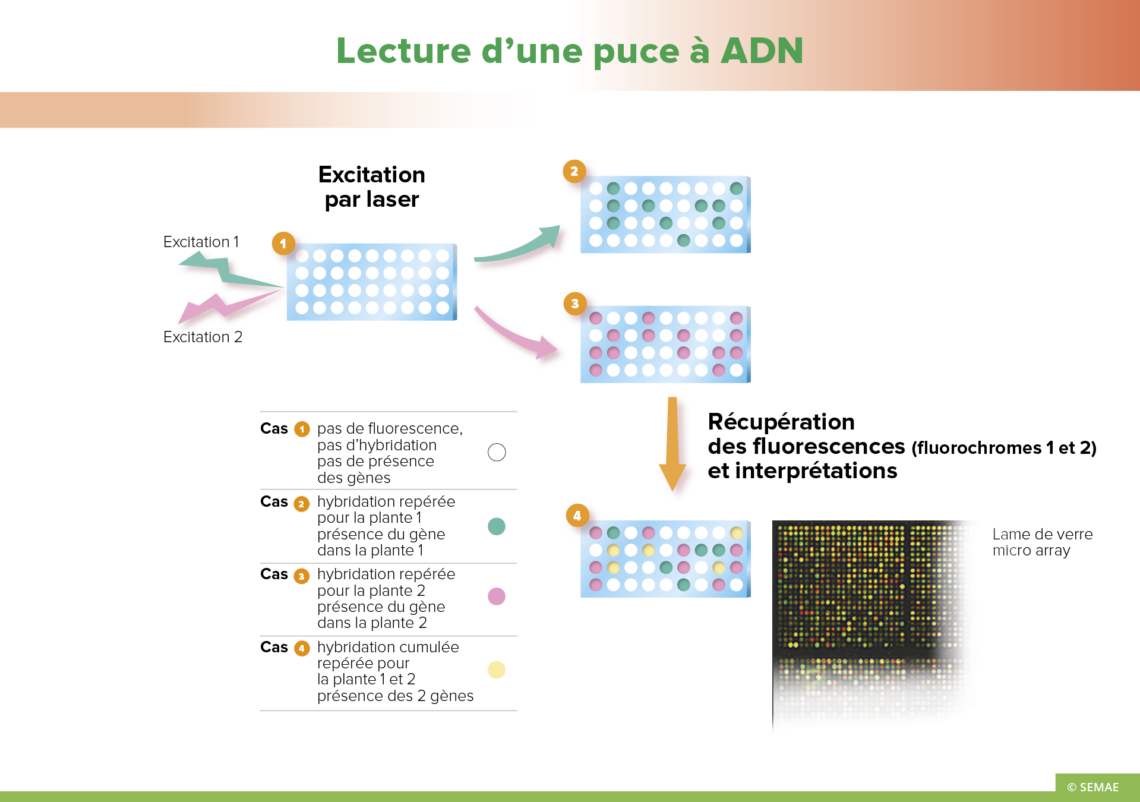
Les puces à ADN sont des images fonctionnelles du génome. Avec l’aide de plusieurs milliers de sondes, il est possible de comparer finement deux plantes. Après excitation laser des lames de verre sur lesquelles les échantillons des plantes ont été déposés, l’interprétation des couleurs de puits permet de déceler les différences génétiques de deux plantes.
Des marqueurs de type RFLP permettent eux aussi de différencier et de caractériser « en routine » des variétés sur un grand nombre de caractères agronomiques.
Les applications
Les puces à ADN permettent d’étudier le transcriptome, c’est-à-dire l’expression des séquences codantes des gènes transcrits en ARN messagers (simple brin). C’est une image fonctionnel du génome, puisque les biopuces ou puces à ADN vont révéler les séquences codantes du génome effectivement exprimées.
La technique repose sur l’hybridation de deux fragments d’acides nucléiques complémentaires ou leur dissociation sous l’action de la température et de la concentration en sel du milieu.
Fabrication des puces à ADN
Une puce à ADN est un support rigide (verre ou nylon) de petite taille sur lequel sont fixés de courtes séquences d’ADN, appelées sondes qui correspondent à des fragments spécifiques (uniques) d’un seul gène. L’ensemble des gènes étudiés sont idéalement déposés sur la puce et chaque position est connue. Les sondes ne sont pas marquées.
Les cibles correspondent aux ARN messagers que l’on souhaite étudier. Pour cela, après extraction des ARN messagers, les chercheurs effectuent une transcription réverse pour obtenir des ADN complémentaires qui sont marqués avec des fluorochromes (ou par des marqueurs radioactifs).
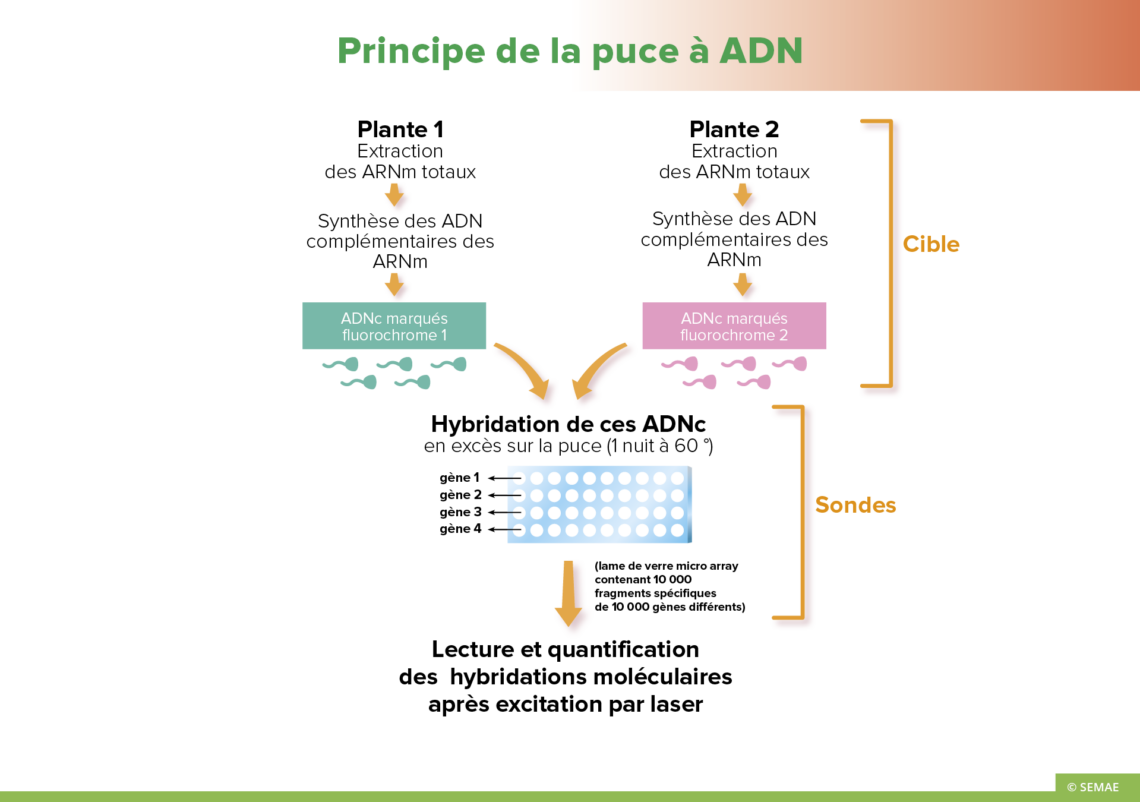
Interaction entre la sonde et la cible
Elle se fait en 12 heures à 60° C environ par interaction des deux chaines de séquences complémentaires qui correspond à une hybridation moléculaire. Si le nombre de sondes sur la puce est bien supérieur aux nombres de cibles (venant s’hybrider sur la puce), il est possible de quantifier les transcrits présents, sachant que le signal de détection est proportionnel à la concentration de la cible.
S’il n’y a pas de signal, c’est que l’hybridation moléculaire n’a pas eu lieu.
Utilisation des puces à ADN
Les puces à ADN permettent de regrouper des gènes ayant le même profil d’expression dans des conditions expérimentales particulières. Elles peuvent par exemple être un outil performant pour l’étude des facteurs abiotiques comme la tolérance à la sécheresse qui impliquent de nombreuses chaines métaboliques et donc de nombreux gènes, car plusieurs milliers, voire plusieurs millions de substitutions de nucléotides peuvent être ainsi étudiées simultanément.
Les différentes puces à ADN
Les filtres à haute densité (macroarrays) sont des plaques en nylon (12X8 cm) qui permettent de quantifier la présence de 2400 gènes par marquage radioactif d’un échantillon (ARNm totaux à étudier).
Les lames de verre (microarrays) permettent de quantifier la présence de 10 000 gènes par marquage fluorescent avec deux conditions expérimentales par lame (voir infographie).
Les puces à oligonucléotides (puce de 1,28cm x 1,28cm) contiennent 300 000 oligonucléotides (sondes) par lame et permettent de quantifier la présence d’ARNm grâce au marquage fluorescent de ces cibles issues d’une seule condition expérimentale.